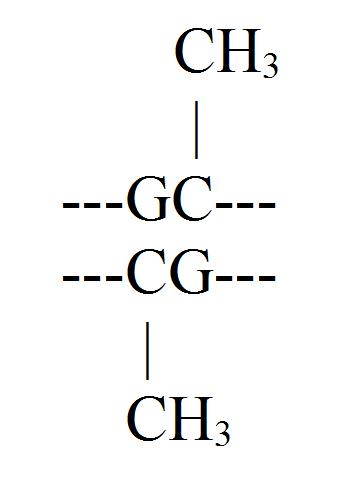4. Unerklärliche Beobachtungen?
5. Epigenetische Mechanismen
6. Erklärung des Unerklärlichen
7. Laborversuche zur Beeinflussung epigenetischer Mechanismen
5. Epigenetische Mechanismen
Mit Hilfe verschiedener Versuche kamen Wissenschaftler der Epigenetik im Labor auf die Spur und entdeckten nach und nach mehrere Mechanismen, welche meist im Zellkern ansetzten und von dort aus einen großen Einfluss auf die biochemischen Prozesse in der Zelle hatten.
5.1 DNA-Methylierung
Einer der bekanntesten epigenetischen Mechanismen, der Einfluss auf die Biochemie einer Zelle hat, führt über die Anlagerung von Methylgruppen an die DNA (Jaenisch et al. 2003). Diese Methylgruppen binden sich allerdings nicht an jede Base der DNA. Bei Wirbeltieren, wie der Mensch eines ist, können sich Methylgruppen nur an die Base Cytosin anlagern und das auch nur, wenn auf die Base Guanin die Base Cytosin folgt.
Da Guanin sich immer an Cytosin bindet, liegt im komplementären Strang die selbe Basenfolge vor und in der Regel ist das Cytosin entweder auf beiden Strängen methyliert, oder auf beiden Strängen unmethyliert.
Am einfachsten lässt sich mit Hilfe von Restriktionsenzymen feststellen, an welchen Stellen sich Methylgruppen an die DNA angelagert haben. Jeder Typ eines Restriktionsenzyms ist in der Lage, DNA bei Vorliegen einer bestimmten Basensequenz zu schneiden. Viele Restriktionsenzyme sind jedoch methylgruppensensitiv, was bedeutet, dass sie nicht schneiden, wenn die DNA an der entsprechenden Stelle methyliert ist. Schneidet man nun 2 Kopien der selben DNA einmal mit methylgruppensensitiven Restriktionsenzymen und einmal mit Restriktionsenzymen, die nicht anfällig für eine Störung durch Methylgruppen sind, so erhält man zwei unterschiedliche Ergebnisse und kann Rückschlüsse auf die methylierten Basen in der DNA ziehen.
Hier zeigt sich bereits eine Auswirkung der Methylgruppen. Bestimmte Enzyme können nicht an DNA binden, die methyliert ist.
Die hier genannten Restriktionsenzyme kommen vor allem bei Bakterien vor. Genutzt werden diese Restriktionsenzyme von bestimmten Bakterien, um DNA zu erkennen, welche nicht ihre eigene DNA ist. Die Enzyme können fremde DNA, wie zum Beispiel die von Viren, zerschneiden, da diese unmethyliert ist. Die Methylierung der eigenen DNA verhindert jedoch, dass das Erbgut der Bakterien Ziel der Restriktionsenzyme wird.
In höheren Lebewesen hat die Methylierung weiterhin eine Schutzfunktion, welche jedoch auf andere Weise funktioniert.
Die Methylgruppen haben Einfluss auf verschiedene Proteine und Proteinkomplexe. Diese Proteinkomplexe werden als Histone bezeichnet. Diese verpacken und komprimieren die DNA und sind Hauptbestandteil des Chromatins. Ist die Anzahl der Methylgruppen an einer Stelle der DNA erhöht, führt das zu einer besseren Komprimierung der DNA an dieser Stelle, so dass es für Enzyme schwieriger ist, an diese Stelle der DNA anzudocken und ihrer Funktion nachzugehen. Das bedeutet im Fall eines DNA-Bereiches, welcher Gene enthält, dass durch die Methylierung der DNA die Aktivität der Gene und dadurch die Menge an produzierter mRNA und produziertem Genprodukt reguliert werden kann. Die Methylierung ist dabei nicht der eigentliche inaktivierende Faktor, sondern verstärkt nur die auf Ebene der Histone angelegte Inaktivierung bestimmter DNA-Abschnitte.
Wo nun liegt die Schutzfunktion? Im Erbgut existieren viele sogenannte Transposons, welche mobile Elemente darstellen, die im Erbgut hin und her springen können. Würden die Zellen diese Transposons nicht regulieren, würde es wiederholt zu unkontrollierten Sprüngen kommen, was ein enormes Risiko für das Erbgut darstellen würde. Methylgruppen binden nun genau im Bereich dieser mobilen Elemente und inaktivieren sie.
Im Bereich der Gentechnik hat die Methylierung durch diese Schutzfunktion Probleme verursacht. Beim Versuch, Gene in die Genome verschiedener Organismen einzuschleusen, stieß man immer wieder auf das Phänomen, dass nach einigen Generationen das eingeschleuste Genprodukt nicht mehr nachweisbar war. Zunächst vermutete man, dass die Gene verloren gegangen seien. Bei genauerer Untersuchung stellte man jedoch fest, dass die entsprechenden Gene weiterhin vorhanden waren, allerdings in methyliertem und damit inaktiviertem Zustand.
Man bezeichnet dieses Stummschalten der Gene, da es vor der Transkription der DNA in mRNA stattfindet, als transkriptionelles Gen-Silencing.
Zusätzlich hat die Methylierung bei Eukaryoten natürlich auch eine regulierende Funktion im restlichen Genom.
Das Methylierungsmuster einer Zelle kann bei der Mitose auf die Tochterzelle weitervererbt werden. Bei der Duplikation der DNA liegt der neu gebildete Strang zwar zunächst in unmethylierter Form vor, eine DNA-Methyltransferase sorgt jedoch dafür, dass die Methylierungsmuster mit dem komplementären Strang abgeglichen werden und die Methylierung weiter vererbt wird. Auf diese Weise können Gewebe trotz Wachstum ein weitgehend gemeinsames Methylierungsmuster beibehalten.
Auch bei der Meiose können die Methylgruppen weiter gegeben werden, es kommt jedoch im Verlauf der Entwicklung eines Organismus mehrfach zu einer starken Veränderung des Methylierungsmusters. Bereits bei der Bildung von Eizellen und Spermien findet eine dieser Veränderungen statt. So werden alte Methylgruppen entfernt und an anderer Stelle neue gesetzt. Vor der Einnistung des Embryos in die Gebärmutterschleimhaut findet eine weitere besonders starke Demethylierung statt, bei der über die Hälfte der von den Eltern geerbten Methylgruppen entfernt werden. Nach der Einnistung werden dann wieder nach und nach Methylgruppen angelagert, bis, beim Menschen ungefähr in der neunten Schwangerschaftswoche das Methylierungsmuster des erwachsenen Organismus erreicht ist.
Die Inaktivierung durch Methylgruppen dient in diesem Zusammenhang als eine Art zelluläres Gedächtnis. Während der Entwicklung müssen zu spezifischen Zeitpunkten Gene aktiv sein und im richtigen Moment wieder inaktiviert werden. Die Methylgruppen führen zu einer solchen Regulation und werden an die nächste Zellgeneration weitergegeben. So bleiben die Gene weiterhin inaktiv und es können sich verschiedene Gewebe ausbilden, welche jeweils ein spezifisches Methylierungsmuster besitzen.
Außerdem können Eltern über diese Methylgruppen Informationen über die zu erwartenden Umwelteinflüsse in Form von Methylgruppen in den Geschlechtszellen speichern und so den Stoffwechsel des Nachwuchses prägen. Besonders anfällig für diese Prägung haben sich zwei spezifische Zeitperioden in der Entwicklung erwiesen.
Zum einen die Zeit im Mutterleib, bei der das noch ungeborene Kind sein individuelles Methylierungsmuster erhält und zum anderen der Lebensabschnitt, in dem die Geschlechtszellen gebildet werden (Okano et al. 1999).
Diese Prägung des Stoffwechsels soll den Nachwuchs auf die entsprechenden Umwelteinflüsse vorbereiten und ist von evolutionärem Vorteil, kann jedoch, bei einer starken Abweichung der eingeprägten von den tatsächlichen Umwelteinflüssen, auch nachteilig für den Nachwuchs sein.
5.2 Die X-Inaktivierung

Abb.4: Barr-Körperchen, Xa(aktiviert) & Xi(inaktiviert)
Foto: Steffen Dietzel
Lizenz: Creative Commons Attribution-Share Alike 3.0 Unported
Die Originaldatei ist hier zu finden.
Methylierung kann, wie bereits erwähnt, im Wirbeltiergenom nur an Stellen stattfinden, an denen eine Guaninbase auf eine Cytosinbase folgt.
An vielen Abschnitten des Genoms kommen diese CG-Sequenzen jedoch besonders oft vor. Man spricht in diesem Zusammenhang von CpG-Inseln. Diese befinden sich oft, aber nicht ausschließlich in den Promotorregionen von Genen, welche essentiell für den Stoffwechsel sind und daher immer aktiv bleiben. Und obwohl diese CpG-Inseln potentielle Bindungsstellen für Methylgruppen darstellen, sind gerade hier in der Regel keine Methylgruppen angelagert.
Zu dieser Regel gibt es jedoch eine Ausnahme. Eines der beiden X-Chromosomen bei Frauen ist in diesen CpG-Inseln stark methyliert und in der Konsequenz, welche wir dem Mechanismus der Methylierung entnehmen, ist dieses X-Chromosom besonders stark komprimiert und weitgehend inaktiv, auch und gerade weil mit Hilfe der Methylgruppen die Promotoren der Gene blockiert werden.
Das Chromatin des einen X-Chromosoms wird dabei so stark komprimiert, dass es bei Anfärbung des Chromatins des Zellkerns als Struktur nachgewiesen werden kann.
Man spricht in diesem Zusammenhang vom sogenannten Barr-Körperchen.
Der Sinn dieser Inaktivierung des einen X-Chromosoms bei Frauen ist die sogenannte Dosiskompensation (Payer et al. 2008).
Da bei Säugetieren das Geschlecht durch die Geschlechtschromosomen X und Y bestimmt wird und das X-Chromosom Gene enthält, die essentiell für den Stoffwechsel sind, das Y-Chromosom jedoch nicht, muss, um die Stabilität des Stoffwechsels zu gewährleisten, die Genaktivität der X-Chromosomen an die der Autosomen angepasst sein. Würden weibliche Zellen mit ihren zwei X-Chromosomen mehr der entsprechenden Genprodukte produzieren als männliche Zellen mit einem X und einem Y-Chromosom, könnte das zu einer gefährlichen Ungleichverteilung der Genprodukte bei einem der Geschlechter führen.
Um diese Ungleichverteilung auszugleichen, wird eines der weiblichen X-Chromosomen inaktiviert.

Abb.5: Schildpattmuster bei einer weiblichen Katze als Beispiel für Zellmosaikbildung
Foto: Ksmith4f
Lizenz: gemeinfrei
Die Originaldatei ist hier zu finden.
Bei anderen Organismen haben sich andere Mechanismen entwickelt, um eine Dosiskompensation zu erreichen. Auch bei diesen besitzen männliche Tiere nur ein X-Chromosom, weibliche jedoch zwei. So reduziert der Fadenwurm C.elegans die Produktion an Genprodukten auf beiden X-Chromosomen des Weibchens um jeweils die Hälfte, während die Taufliege Drosophila melanogaster die Produktion an Genprodukten auf dem einen X-Chromosom des männlichen Tiers auf das doppelte erhöht.
Dass sich gleich 3 verschiedene Mechanismen entwickelt haben, um diese Dosiskompensation zu erreichen, ist auch ein Indiz dafür, wie wichtig diese evolutionär für die Stabilität des Stoffwechsels ist.
Bei Säugetieren bleibt nun die Frage, welches der X-Chromosomen zu einem Barr-Körperchen wird und welches nicht.
Bei den Beuteltieren wird das X-Chromosom des Vaters inaktiviert. Bei allen anderen Säugetieren ist das zunächst noch genauso, doch im Entwicklungsstadium der Morula wird diese Inaktivierung wieder rückgängig gemacht und es kommt im Blastocystenstadium erneut zu einer Stilllegung eines der X-Chromosomen. Welches der X-Chromosomen in diesem Stadium inaktiviert wird, ist in jeder einzelnen Zelle der Blastocyste jedoch vom Zufall bestimmt und bleibt für alle Tochterzellen bestehen.
Aus diesem Grund kann man Frauen als Zellmosaike bezeichnen, da je nach Abstammung der Zellen eines Gewebes das eine oder das andere X-Chromosom aktiv ist.
5.3 Modifikation von Histonen
Histone sind, wie bereits erwähnt, Proteine, die im Zellkern für die Verpackung der DNA sorgen und damit auch regulatorische Aufgaben übernehmen, da die Expression von Genen durch Bindung der DNA an die Histone reguliert werden kann (Goll et al. 2002).
Lange Zeit wurde den Histonen nur eine Funktion als Packmaterial zugeschrieben, ihre epigenetischen Funktionen sind erst in den letzten zwei Jahrzehnten erkannt worden.
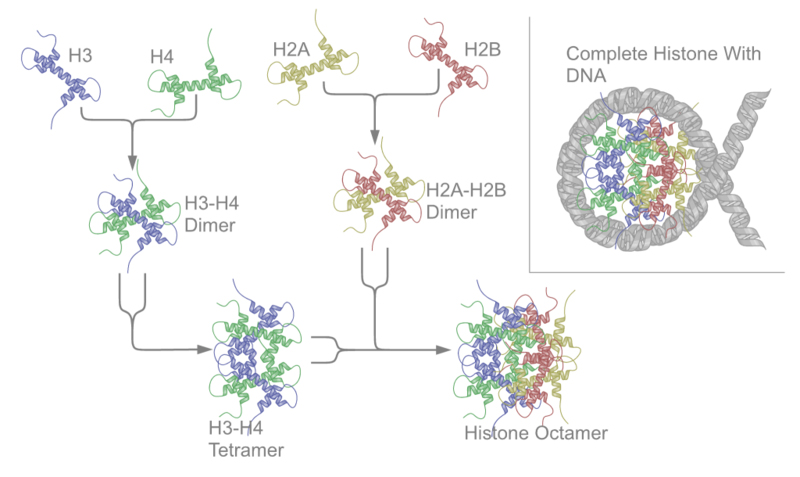
Abb.6: Aufbau eines Nukleosoms aus Histonen
Foto: Richard Wheeler (Zephyris)
Lizenz: Creative Commons Namensnennung-Weitergabe unter gleichen Bedingungen 3.0 Unported
Die Originaldatei ist hier zu finden.
Es sind 5 verschiedene Haupt-Histon-Proteine bekannt. Sie werden als H1, H2A, H2B, H3 und H4 bezeichnet. Jeweils 2 Moleküle von H2A, H2B, H3 und H4 bilden gemeinsam den Kern eines Nukleosoms, auf welches die DNA aufgewickelt ist. Schematisch ähnelt das einer Perlenschnur.
Das Histon H1 ist an der Stelle zu finden, an der sich die DNA um das Histon herum windet und wird daher mit der Befestigung der DNA an den Nukleosomen in Verbindung gebracht. Genau ist die Funktion des Histons H1 jedoch nicht geklärt.
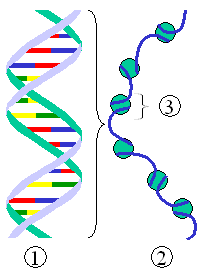
Abb.7: Packung der DNA durch Nukleosome
Foto: Joost de Jong
Lizenz: Creative Commons Attribution-Share Alike 3.0 Unported
Die Originaldatei ist hier zu finden.
Nukleosomen besitzen aufgrund der an ihnen beteiligten Histone eine Präferenz für bestimmte Bereiche der DNA, womit die Basenfolge der DNA bis zu einem bestimmten Grad Einfluss darauf hat, an welchen Stellen Nukleosome angelagert werden. Damit existiert ein Einfluss auf das Verteilungsmuster der Nukleosomen an der DNA. Die Basenfolge bestimmt also bis zu einem gewissen Grad mit, welche Bereiche des Chromatins wie stark kondensiert sind.
Die Enden der Histonmoleküle ragen als Histon-Schwänze aus den Nukleosomen heraus. Am N-terminalen Ende eines Histons können verschiedene Enzyme angreifen und unterschiedliche Modifikationen vornehmen.
Diese Modifikationen wirken sich auf die Struktur der Histone aus und ändern auf diese Weise unter anderem den Grad der Kondensation des Chromatins, was wiederum einen direkten Einfluss auf das Ablesen von Genen in den betroffenen Bereichen des Chromatins hat. Durch die unterschiedliche Kondensation kann das Chromatin in Heterochromatin und Euchromatin unterschieden werden. Heterochromatin bezeichnet dabei das hoch kondensierte und stark verdichtete Chromatin, in dem kaum Genaktivität herrscht. Als Euchromatin bezeichnet man demnach das weniger dicht gepackte Chromatin, in dem hohe Genaktivität herrscht.
Bei der Modifikation der Histon-Schwänze handelt es sich um verschiedene chemische Veränderungen. Die Auswirkung einer Modifikation hängt von ihrem Typ ab, so wird zum Beispiel durch eine Phosphorylierung eines Histon-Schwanzes der Zugang zur DNA und der auf ihr liegenden Gene erleichtert, während die auch bei der DNA genutzte Methylierung eines Histon-Schwanzes den Kondensationsgrad des Chromatins erhöht und damit den Zugang erschwert.
Wie bereits beschrieben werden die Methylgruppen, welche an die DNA angelagert sind, in verschiedenen Stadien der Entwicklung eines Organismus umorganisiert oder entfernt. Wie dennoch eine Vererbung von Methylgruppen stattfinden kann, ist teilweise noch unbekannt. Man vermutet jedoch, dass auf der Ebene der Histone eine entsprechende Vererbung stattfinden könnte, die daraufhin wieder zu einer Anlagerung von Methylgruppen an die DNA führen würde.
5.4 RNA-Interferenz
Einen beziehungsweise mehrere weitere epigenetische Mechanismen bildet die RNA-Interferenz. Entdeckt wurde die RNA-Interferenz mehr oder weniger zufällig bei verschiedenen genetischen Versuchen, bei denen, wie auch schon bei der DNA-Methylierung beschrieben, Gene inaktiviert wurden.
Unter anderem wurde versucht, eine intensivere Färbung bei Petunien zu erreichen und den Petunien daher eine zweite Kopie des Gens eingepflanzt, welches für den blauroten Blütenfarbstoff verantwortlich ist (Napoli et al. 1990).

Abb.8: RNA-Interferenz bei Petunien
links: Wildtyp
Mitte und rechts: transgene Blüten
Foto: Marjori A. Matzke, Antonius J. M. Matzke; credit Jan Kooter for the left and middle images, and Natalie Doetsch and Rich Jorgensen for the right images
Lizenz: Creative Commons Attribution 2.5 Generic
Die Originaldatei ist hier zu finden.
Dabei entsprach das Ergebnis nicht den Erwartungen, da die Blüten überraschenderweise keine stärkere Färbung aufwiesen, sondern stattdessen weiße Flecken entwickelten oder sogar völlig farblos blieben. Bei genauerer Untersuchung entdeckte man, dass nicht nur die Gene verstummt waren, sondern gleichzeitig die bereits vorhandene mRNA abgebaut wurde.
Bereits zuvor war bekannt, dass man ähnliche Ergebnisse mit einer sogenannten antisense-RNA erreichen konnte. Die antisense-RNA stellt dabei eine komplementäre Kopie der mRNA dar.
Zunächst war der dahinter verborgene Mechanismus natürlich unbekannt, dennoch wurde er genutzt, indem man gezielt eine antisense Version eines Gens in Zellen implantierte. Auf diese Weise schaltete man das eigentliche Gen aus und anhand der Auswirkungen auf den Phaenotyp konnte auf die Funktion des Gens geschlossen werden.
Der nächste Schritt war die Entdeckung, dass der rätselhafte Mechanismus noch besser funktionierte, wenn man gezielt doppelsträngige RNA in die Zellen einschleuste. Als man daraufhin kleine RNA-Fragmente entdeckte, welche von der eingeschleusten RNA zu stammen schienen, entdeckte man den dahinter liegenden Mechanismus, welcher der zellulären Abwehr diente.
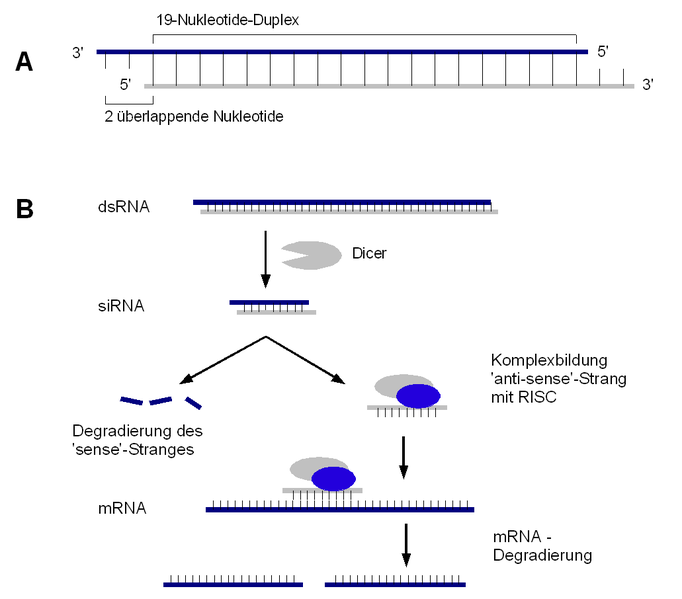
Abb.8: Mechanismus der RNA-Interferenz
Foto: PhilippN
Lizenz: Creative Commons Namensnennung-Weitergabe unter gleichen Bedingungen 2.5 US-amerikanisch (nicht portiert)
Die Originaldatei ist hier zu finden.
Bei diesem Mechanismus spielt zunächst ein Enzym mit der Bezeichnung DICER eine Rolle, welches gezielt doppelsträngige RNA in 21 bis 24 Basenpaare lange Stücke zerlegt.
Diese Fragmente bezeichnet man als small-interfering-RNAs oder abgekürzt siRNAs. Diese kleinen RNA-Fragmente verbinden sich mit mehreren weiteren Proteinen, welche gemeinsam den sogenannten RISC Enzymkomplex bilden (Bagasra et al. 2004). Dabei wird nur der antisense-Strang des doppelsträngigen RNA-Fragments mit in den Enzymkomplex eingebaut.
Mit Hilfe dieser RNA ist der RISC-Komplex nun in der Lage, gezielt nach RNA zu suchen, welche zu der Basenfolge der in ihn eingebauten RNA passt. Wenn ein passendes Fragment gefunden wurde, so wird es von RISC zerschnitten und damit unbrauchbar gemacht (siehe Abb.8).
Da viele Viren und auch Transposons zumindest zeitweilig in ihrer Entwicklung doppelsträngige RNA produzieren, kann dieser Verteidigungsmechanismus hier besonders gezielt eingesetzt werden. Viren werden gezielt bekämpft und bei Transposons wird ein Springen verhindert. Außerdem bleibt der RISC-Komplex auch nach dem Erfüllen seiner Aufgabe funktionstüchtig und kann eine gewisse Resistenz verleihen. Bei Zellteilungen kann durch die Aufteilung des Zellplasmas unter den Tochterzellen außerdem auch eine Aufteilung der vorhandenen Enzymkomplexe unter den Tochterzellen stattfinden und damit eine gewisse Resistenz aufrecht erhalten und vererbt werden.
Dieser Verteidigungsmechanismus hat jedoch im Laufe der Evolution eine weitere Aufgabe erhalten und dient nun auch der Regulation von Genen. Dazu werden von der Zelle gezielt kleine RNAs, sogenannte microRNAs, produziert, welche durch Faltung doppelsträngig werden. Auch sie werden von einem DICER zerteilt und auch ihre Fragmente verbinden sich mit Proteinen zu einem RISC-Komplex.
Die microRNA führt dazu, dass der RISC-Komplex nun für eine bestimmte mRNA spezifisch ist, diese zerschneidet und so die Proteinproduktion stoppt. Da die microRNA aber nicht immer genau an die mRNA passt, kommt es auch häufig dazu, dass der RISC-Komplex an unpassende, aber sehr ähnliche mRNA andockt, welche er dann nicht zerschneiden kann. Während dieser Komplex an der mRNA angedockt ist, kann jedoch keine Translation der entsprechenden mRNA stattfinden. Auf diese Weise kommt es zu einer Behinderung der Translation der mRNA in Proteine, wodurch der RISC-Komplex zu einem Werkzeug wird, mit dem die Zelle ihre Proteinsynthese genau und effizient regulieren kann.
Es handelt sich damit um eine gezielte Regulation und Stilllegung der Gene nach der Transkription und der Prozess wird daher als post-transkriptionelles Gen-Silencing bezeichnet.
Ein weiterer bei Pflanzen entdeckter Bereich der RNA-Interferenz bildet Schnittstellen zu den bereits genannten Mechanismen des transkriptionellen Gen-Silencing durch Modifikation von Histonen und DNA (Aufsatz et al. 2002). Der fragliche Mechanismus ist mit Hilfe kleiner RNA-Fragmente dazu in der Lage, spezifisch passende DNA-Sequenzen zu methylieren oder Histon-Schwänze zu modifizieren. Es handelt sich also um eine weitere Steuerungsmöglichkeit des transkriptionellen Gen-Silencing. Inwieweit dieser Mechanismus auch bei anderen Organismen vorkommt, ist noch weitgehend ungeklärt, doch es häufen sich die Indizien, dass dieser Mechanismus weit verbreitet ist.
Dieser Mechanismus könnte auch erklären, auf welchem Weg Umwelteinflüsse zu einer Prägung, einem sogenannten Imprinting des Nachwuchses führen und wie eine gezielte Methylierung des Genoms vorgenommen werden kann.
vorherige Seite – Gliederung 1 2 3 4 5 6 7 8 9 – nächste Seite